RToate rezultatele preconizate în Planul de realizare al proiectului au fost obținute, după cum urmează:
- Rezultat 1. Raport privind metodologia și protocoalele de lucru folosite în cercetare
Au fost elaborate următoarele documente, ce au fost încărcate pe platforma EVOC, la categoria RST-Rezultate / Studii:
• Metodologie de colectare și analiză a datelor în condiții de laborator
• Protocol implementare, testare, confidențialitatea datelor
• Protocol de cercetare pentru secvențierea tulpinilor SARS-CoV-2
• Protocol analiză bioinformatică variante genomice tulpini SARS-CoV-2 circulante în RO
- Rezultat 2. Pagina web a proiectului - http://rogenofil.usv.ro
- Rezultat 3. Bază electronică de date biologice, clinice şi demografice
A fost completată baza electronică de date biologice, clinice şi demografice proiectată în prima etapă a acestui proiect. Astfel, au fost introduse 1757 seturi de date, reprezentând tot atâtea probe primare, provenite de la partenerii P1-P5, precum şi de la Direcţii de Sănătate Publică din judetele Iaşi, Botoşani şi Neamţ. Baza cuprinde date grupate pe:
• Variabile demografice: vârstă, sex, localitate de provenienţă
• Variabile epidemiologice: călătorie, contacţi (4 coloane)
• Variabile clinice: comorbidităţi, simptome, imagistică, tratament (45 de coloane)
• Variabile biochimice: markeri sanguini (62 de coloane)
Datele au fost folosite pentru analizele statistice din cadrul modelelor factorilor de risc pentru COVID-19. Bază electronică de date biologice, clinice şi demografice se află la sediul USV populată cu datele corespunzătoare celor două etape ale proiectului iar o prezentare sintetică a acesteia a fost încarcată pe platforma EVOC, la categoria RST-Rezultate / Altele, în documentul cu denumirea Bază electronică de date biologice, clinice şi demografice.
- Rezultat 4. Biobancă extracte acizi nucleici
- Rezultat 5. Bază de date publică secvenţe genomice SARS-Cov-2 circulante pe teritoriul României
- Rezultat 6. Lista accesiuni secvențe Sars-CoV-2 arhivate în baze de date internaționale
- Rezultat 7. Raport rezultate analiza filogenetica pe zone geografice a Sars-Cov-2
- Rezultat 8. Model evolutiv al variantelor Sars-CoV-2 pe teritoriul României
- Rezultat 9. Model predictiv privind factori de risc pentru infectarea cu Sars-Cov-2
- Rezultat 10. Raport cu privire la numărul estimat de infectați/recuperați/decese pe teritoriul RO
- Rezultat 11. Raport final rezultate și interpretare
- Rezultat 12. Raport final de activitate proiect
- Rezultat 13. Raport financiar final al proiectului
- Rezultat 14. Raport de audit final al proiectului
-
Înregistrarea pentru utilizarea modelelor și a bazelor de date obținute se face prin crearea unui cont de utilizator în urma contactării directorului de proiect.
Biobanca a fost completată cu 1304 de eluate ARN viral, stocate la -80o într-un ultracongelator aflat la sediul instituției coordonatoare în cadrul Laboratorului de Metagonomică şi Biologie Moleculară. Cele 1304 de eluate provin dintr-un număr total de peste 1757 de probe primare, din care au fost selectate doar cele conforme standardelor de calitate impuse prin protocolul de cercetare pentru primele etape din protocolul de secvenţiere. Pe platforma EVOC au fost încărcate fotografii reprezentative, cu probe constituente ale biobăncii, la categoria RST-Rezultate/Altele, în documentul asociat rezultatului cu denumirea Biobancă extracte acizi nucleici.
Informaţiile asociate secvențelor genomice SARS-CoV-2 circulante pe teritoriul României realizate cadrul proiectului au fost introduse în baza de date a proiectului ce este disponibilă la adresa http://solutiisarscov2.usv.ro:5000/, credenţialele de acces la această platforma publică fiind obţinute pe baza înregistrării online. Au fost introduse rezultatele a 1026 de secvenţieri dintre care: 902 de secvențieri prin tehnologia NGS Ion Torrent, 112 secvenţieri prin tehnologia Sanger şi 12 secvenţieri prin tehnologia Nanopore. Majoritatea secvenţierilor realizate prin tehnologia NGS au condus la secvenţe genomice ce satisfac standarde înalte de calitate şi au fost indexate în Baza de date publică internaţională GISAID ( https://www.gisaid.org/ ). Lista cu secvenţele din baza de date publică a fost încărcată pe platforma EVOC, la categoria RST-Rezultate/Altele, în documentul Bază de date publică secvenţe genomice SARS-CoV-2 circulante pe teritoriul RO.
Peste 70% dintre secvenţierile realizate prin tehnologia NGS au condus la secvenţe genomice ce satisfac standarde înalte de calitate, iar informațiile rezultate din asamblarea, adnotarea și compararea acestor secvențe genomice ale SARS-CoV-2 au fost indexate în Baza de date internaţională GISAID - Global initiative on sharing all influenza data https://www.gisaid.org/. Astfel, un număr de 603 de secvenţele efectuate în cadrul proiectului prin tehnologia NGS se află indexate în GISAID. Majoritatea secvenţelor rezultate din proiect încărcate în GISAID s-au încadrat la categoria „High quality”, fiind secvenţe cu peste 27000 de nucleotide, fără regiuni semnificative de tip „NNN” şi fără artefacte semnificative. Aceasta lista poate servi pentru analize filogenetice ulterioare, din diverse regiuni ale României, iar secvențele propriu-zise și metadatele asociate pot fi ușor obținute pe baza indicativului EPI GISAID. Secvențele genomice au fost incluse și în platforma publică Nextstrain, https://nextstrain.org/, unde fac parte din analizele filogenetice generate automat, la nivel mondial. Lista cu secvențele arhivate în GISAID a fost încărcată pe platforma EVOC, la categoria RST-Rezultate / Altele, în documentul Lista accesiuni secvențe Sars-Cov-2 arhivate în baze de date internaționale.
Folosind modelul longitudinal al evoluţiei variantelor SARS-CoV-2 pe teritoriul României, dezvoltat în cadrul proiectului, a fost realizată analiza filogenetică pe zone geografice a SARS-CoV-2 fiind descrise posibilele rute și date de introducere pentru variantele principale SARS-CoV-2 în fiecare din regiunile de dezvoltare ale României, precum şi posibilele relații filogenetice între variantele detectate. Echipa proiectului din cadrul instituţiei coordonatoare a implementat o platformă electronică de analiză filogeografică şi de vizualizare a rezultatelor, care cuprinde majoritatea secvenţelor SARS-CoV-2 circulante pe teritoriul României disponibile în baza de date internaţională GISAID, precum şi secvenţe contextuale de la nivel global decărcate din aceeaşi bază GISAID. Astfel, platforma conţine 11099 secvenţe SARS-CoV-2, grupate şi analizate la nivel naţional sau pe regiuni geografice, 7562 dintre aceste secvenţe fiind de pe teritoriul Romaniei. Într-o primă fază s-a calculat incidenţa principalelor variante virale circulante pe teritoriul României, în funcţie de zona geografică. Distribuţia principalelor variante virale în funcţie de regiunea geografică şi de data eşantionării este ilustrată în Figura 1 (axa orizontală: regiunea de dezvoltare; axa verticală: data eşantionarii; fiecare culoare indicând o anumită variantă SARS-CoV-2). Deşi la nivel naţional SARS-CoV-2 a avut o evoluţie similară cu cea de la nivel global, în cadrul diferitelor regiuni au existat anumite particularităţi de răspândire. Acestea, cel mai probabil, au fost influenţate de factori socio-demografici: mobilitatea diferită a populaţiei, gradul diferit de respectare a restricţiilor, rata diferită de vaccinare. Posibilele rute de transmitere a SARS-CoV-2 pe teritoriul României, estimate pe baza disponibilităţii genomurile secvenţiate, sunt prezentate în Figura 2.
S-a constatat că posibilele surse de introducere a diverselor variante ale virusului SARS-CoV-2 în România, şi pe regiuni, sunt ţări precum Italia, Marea Britanie, Cipru, ceea ce corespunde cu anumite tipare de mobilităţi ale cetăţenilor români. Pe de altă parte, este indicată şi posibilitatea introducerii variantelor din ţări precum Rusia, Belgia, Franţa sau, într-o mai mică masură, chiar de pe continentul american. De asemenea, se disting potenţiale tipare de evoluţie în plan naţional sau regional. O analiză detaliată este prezentată în Raportul încărcat pe platforma EVOC la categoria RST-Rezultate/Studii, în documentul Raport rezultate analiza filogenetică pe zone geografice a Sars-CoV-2.
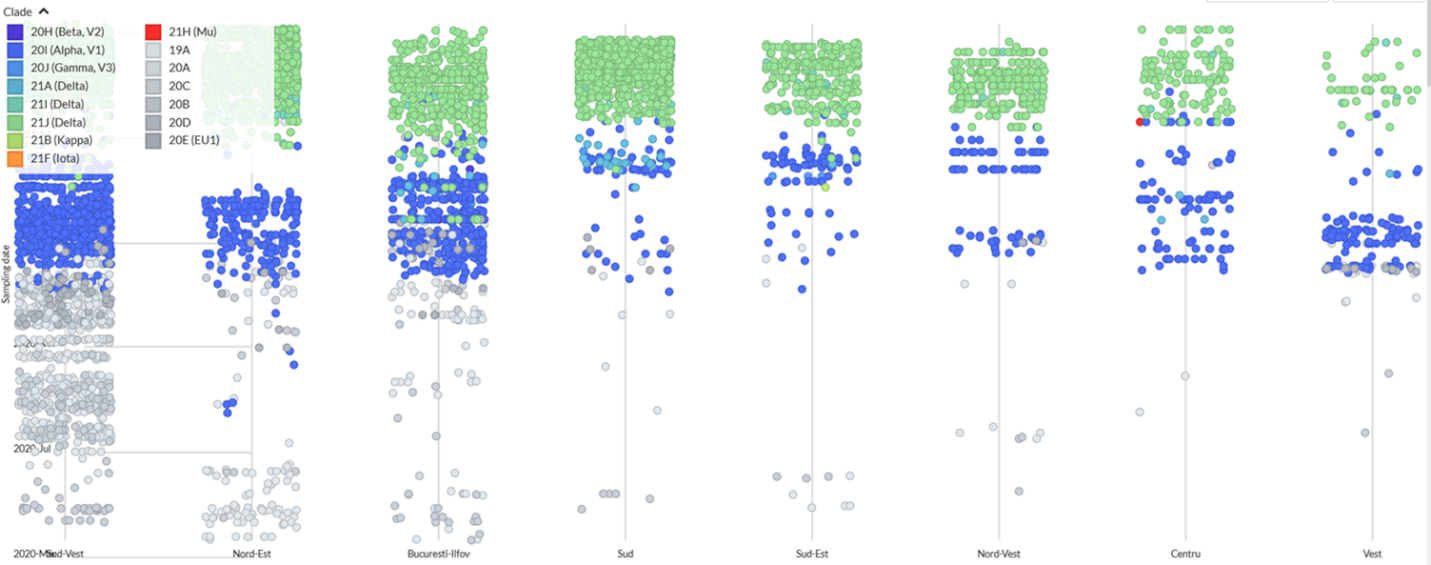
Figura 1. Distribuţia principalelor variante virale, în funcţie de data eşantionării şi de regiunea geografică (axa verticală: data eşantionării; axa orizontală: regiunea de dezvoltare; fiecare culoare indicând o anumită variantă SARS-CoV-2).
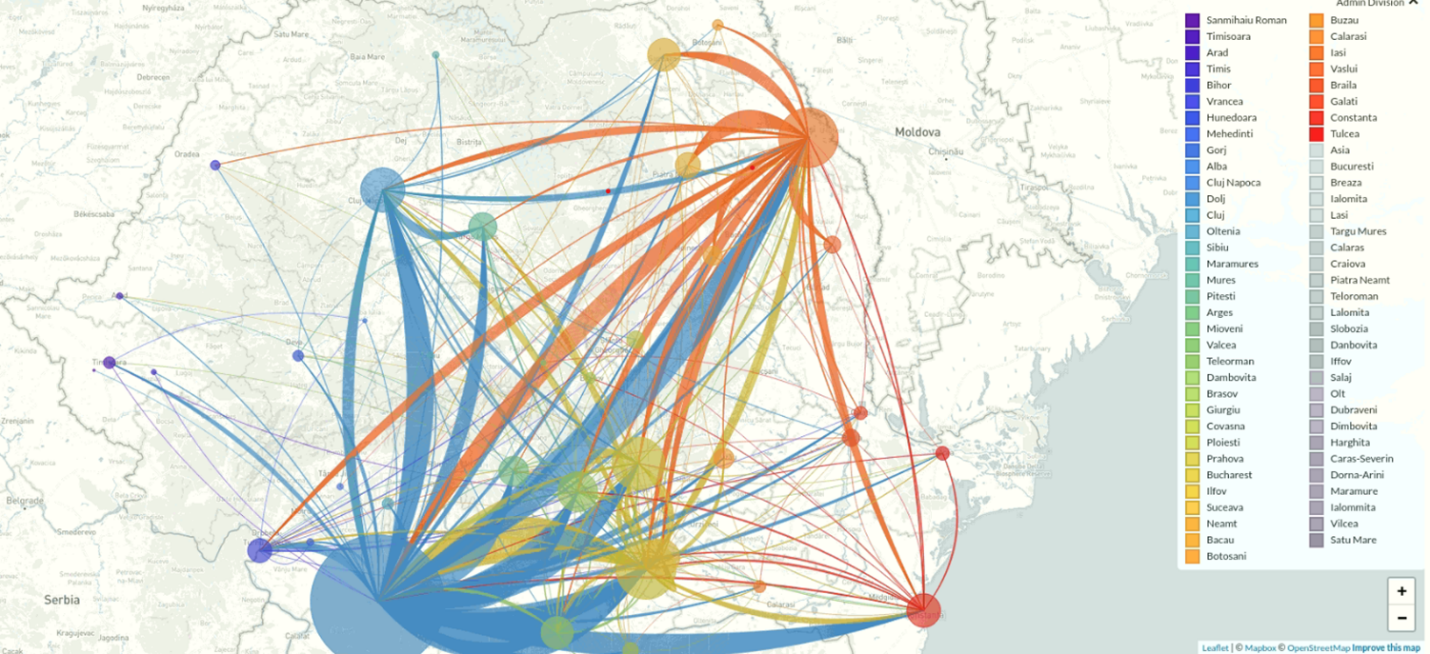
Figura 2. Posibilele rute de transmitere a SARS-CoV-2 pe teritoriul României, estimate pe baza disponibilităţii genomurile secvenţiate
Modelul evolutiv al variantelor SARS-Cov-2 circulante pe teritoriul României a fost dezvoltat şi implementat pe serverele instituţiei coordonatoare, iar interfaţa de vizualizare este disponibilă la http://solutiisarscov2.usv.ro:5000/, credenţialele de acces la platformă fiind obţinute pe baza înregistrării online. Modelul a fost folosit pentru analiza evolutivă la nivel naţional, determinându-se posibilele introduceri ale virusului în ţară şi analizându-se diversitatea şi incidenţa virusului pe regiuni. Raportul de prezentare a modelului şi analiză a rezultatelor la nivel naţional a fost încărcat pe platforma EVOC la categoria RST-Rezultate / Studii, în documentul Model evolutiv al variantelor SARS-CoV-2 pe teritoriul României – Raport analiză date. În ansamblu, România a avut toate tulpinile majore, circulante la nivel global, de la începutul pandemiei – Figura 3, cu posibile variante localizate. În cadrul regiunilor geografice considerate separat, evoluția a fost similară cu cea la nivel naţional, dar cu o reprezentare diferită a genomurilor, în funcție de disponibilitatea recoltării și secvențierii în regiunea respectivă în distribuţia acestor variante.
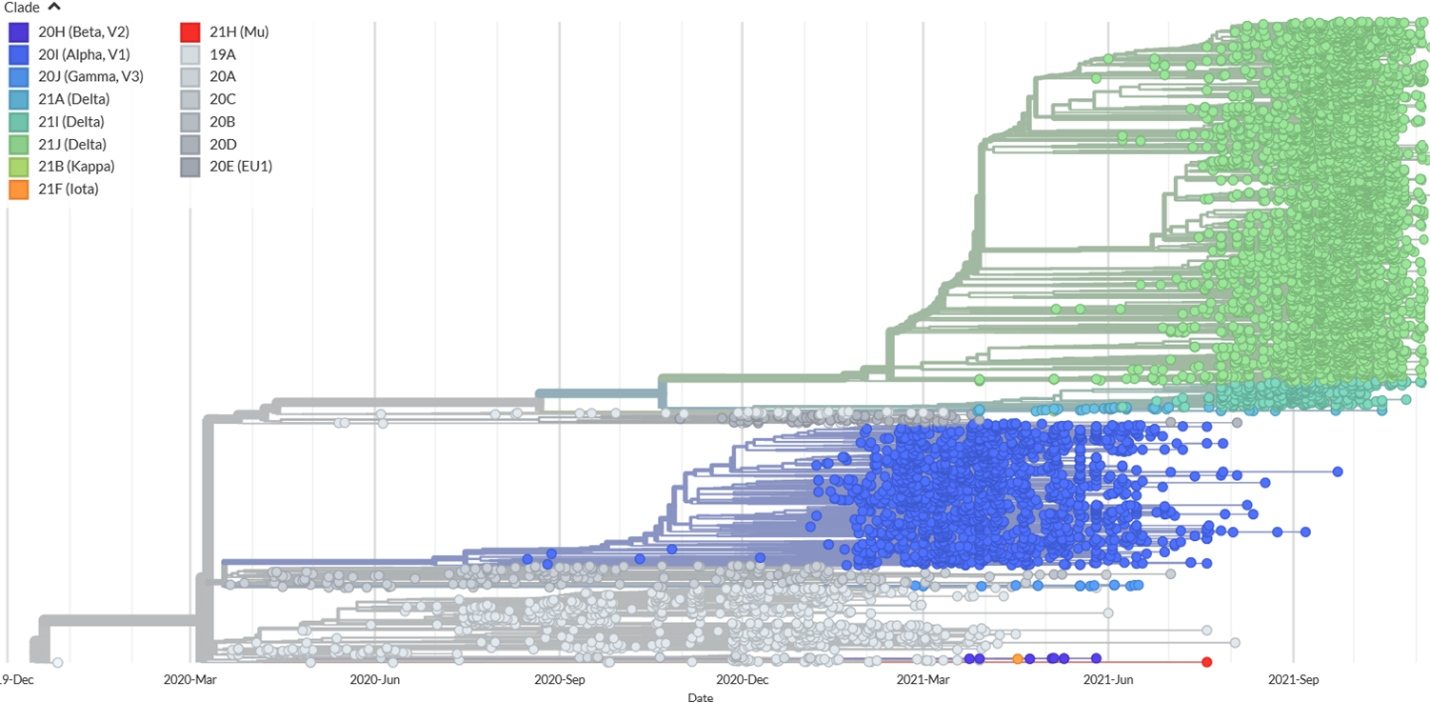
Figura 3. Evoluţia temporală a incidenţei Sars-CoV-2 pe teritoriul României.
Informaţiile clinice, biochimice şi demografice au fost identificate, prelucrate şi evaluate conform ipotezelor predictive şi de asociere privind factorii de risc. Inițial, s-au analizat variabilele descriptive ale seturilor de date şi s-au eliminat valorile extreme. Pentru dezvoltarea modelului predictiv privind factori de risc pentru infectarea cu coronavirus s-au realizat module în limbajul de programare statistică şi mediul software R pentru: determinarea modelului de distribuţie a datelor, în vederea selectării metodelor statistice adecvate; reprezentarea grafică a distribuţiei valorilor, vizualizării și verificării uniformităţii acestora şi a valorilor reziduale, în functie de tipul de variabilă (nominală, ordinală, binară, etc); analize de tip regresie pentru a evidenţia posibilele asocieri între variabile dependente de gazdă, severitatea bolii şi rata de deces.
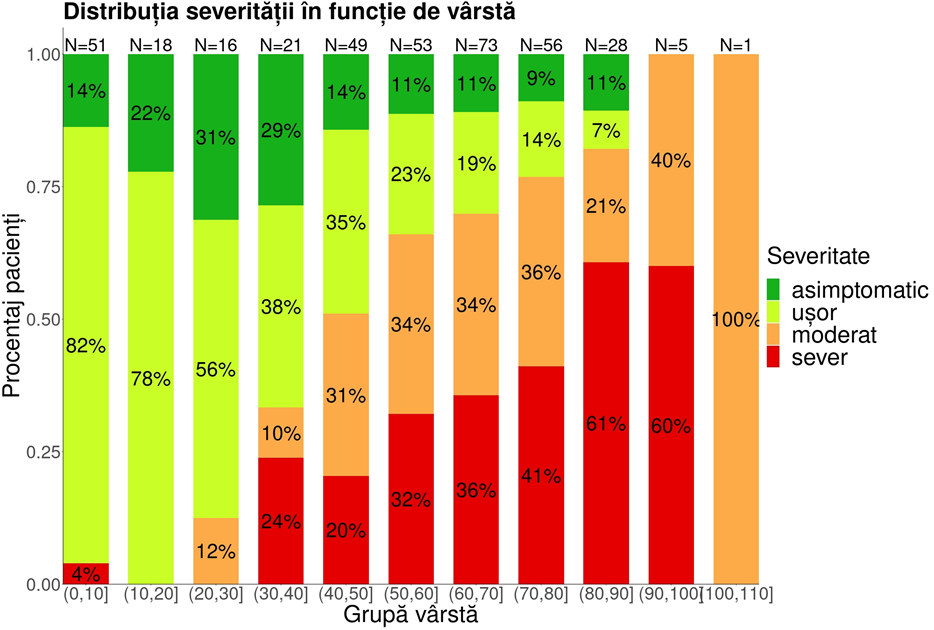
Figura 4. Distribuția severității Covid-19 pe grupe de vârstă la pacienții analizaţi.
De asemenea, s-au implementat modele de analiză de tip regresie şi cluster utilizând platforma software SPSS pentru evaluarea posibilelor asocieri între variabilele dependente de virus (varianta virală şi prezenţa anumitor mutaţii) şi severitatea bolii. Datele obţinute folosind modelul predictiv privind factori de risc pentru infectarea cu SARS-CoV-2 au fost analizate şi interpretate în contextul literaturii de specialitate. În primă fază, s-au comparat valorile descriptive ale fiecărui parametru cu cele descrise în lucrări ştiintifice relevante. Ulterior, s-au identificat posibilele mecanisme care pot explica variaţiile parametrilor consideraţi și s-a analizat semnificaţia pe care valorile obţinute o pot avea, în contextul transmiterii și răspândirii virale, dar și al duratei de spitalizare. Pentru acei parametri identificați ca variabili semnificativ în context COVID-19 s-a calculat rata de risc în conjuncţie cu vârsta, sexul sau comorbidităţile prezente. În mare parte, rezultatele se corelează cu observațiile efectuate la nivel mondial asupra pacienților COVID-19. Raportul de prezentare a modelului şi analiză a rezultatelor a fost încărcat pe platforma EVOC la categoria RST-Rezultate / Studii, în documentul Model predictiv privind factori de risc pentru infectarea cu SARS-Cov-2 – Raport analiză date.
Folosind analizele datelor publicate de autorităţi naţionale şi comparaţii ale evoluţiei pandemiei COVID-19 în diverse ţări, modelul longitudinal al evoluţiei variantelor SARS-CoV-2 pe teritoriul României şi modelul predictiv privind factori de risc pentru infectarea cu SARS-Cov-2, precum şi modele compartimentale de tip SEIR (susceptibili, expuşi, infectaţi, recuperaţi) au fost realizate estimări ale numărului total de persoane infectate/recuperate/ decedate la nivel naţional. Raportul elaborat a fost încărcat pe platforma EVOC la categoria RST-Rezultate/Studii, în documentul asociat rezultatului cu denumirea Raport cu privire la numărul estimat de infectaţi/recuperaţi/decese pe teritoriul României
Au fost elaborate două Rapoarte științifice și tehnice ce au descris activitatea desfășurată şi rezultatele obţinute în cadrul celor două etape ale proiectului și au fost încărcate pe platforma EVOC, la categoria RST-File.
A fost completat prezentul Raportul final de activitate proiect, în conformitate cu formatul transmis de autoritatea contractantă şi a fost încărcat pe platforma EVOC, ca document suplimentar la categoria RST File.
Rapoartele financiare al etapelor I şi II ale proiectului au fost compuse din Fișele de evidență a cheltuielilor pe capitole, Devizele post-calcul 2020 şi 2021 și Acordurile de realocare 2020 şi 2021, documente ce au fost completate pe platforma EVOC și validate de responsabilul tehnic alocat proiectului.
Rapoartele de audit ale etapelor I şi II ale proiectului au fost realizate de un auditor financiar extern și au fost încărcate pe platforma EVOC, la categoria Audit.
În concluzie, toate rezultatele preconizate în Planul de realizare al proiectului au fost obținute.
Chiar dacă proiectul nu a avut drept scop o monitorizare în timp real a variantelor care determinau îngrijorare la nivel european şi naţional, echipa proiectului a răspuns la necesităţile urgente apărute pe plan naţional pe parcursul derulării lui şi a diseminat, cu celeritate, rezultatele în rândul autorităţilor competente prin rapoartele sintetice săptămânale, în perioadele critice, către Ministerul Sănătăţii - Institutul Naţional de Sănătate Publică şi a indexat cu celeritate rezultatele secvenţierilor în principala baza de date internaţională de profil GISAID - Global initiative on sharing all influenza data. Rezultate ale proiectului au fost, de asemenea, diseminate public prin intermediul a mai mult de 100 de intervenţii în mass-media regională, naţională şi chiar internaţională precum și prin intermediul articolelor ştiinţifice: Introduction and Characteristics of SARS-CoV-2 in North-East of Romania During the First COVID-19 Outbreak - publicat în Frontiers in Microbiology (iulie 2021, factor de impact ISI 6,320, cuartila ISI Q1 în domeniul Microbiologie); A Conservative Replacement in the Transmembrane Domain of SARS-CoV-2 ORF7a as a Putative Risk Factor in COVID-19 – publicat în Biology (decembrie 2021, factor de impact ISI 5,079, cuartila Q1 în domeniul Biologie); Emergence of the First Strains of SARS-CoV-2 Lineage B.1.1.7 in Romania: Genomic Analysis – publicat în JMIRx Med (august 2021), alte lucrări cu rezultate obţinute în cadrul proiectului aflându-se în diverse stadii de pregătire pentru publicare.
